Нова технологія прискорить боротьбу з патогенами, які загрожують сільськогосподарським культурам
06.02.2019
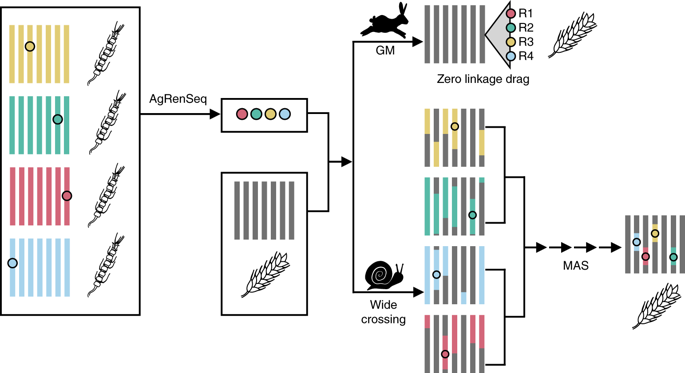
Методика AgRenSeq (прискорене клонування) була розроблена дослідниками Центру Джона Іннеса разом з колегами в США і Австралії, щоб прискорити боротьбу з патогенами, які загрожують харчовим культурам у всьому світі.Дослідники розробили новий метод, який дозволяє їм швидко залучати гени стійкості до хвороб з диких рослин і переносити їх на культурні рослини.
Дослідники можуть використовувати лабораторні методи для клонування генів та впровадження їх у елітні сорти вітчизняних культур, щоб захистити їх від патогенів та шкідників, таких як іржа, борошниста роса та гессенська муха.
AgRenSeq допоможе поліпшити врожаї та зменшити використання пестицидів, говорить д-р Бранд Вульф, керівник проекту в Центрі Джона Іннеса.
“Маючи AgRenSeq в нашому інструментарії означає, що сільськогосподарські культури можуть бути більш стійкими, що означає більш високі врожаї та зниження залежності від пестицидів для захисту культур. Ми знайшли спосіб сканувати геном дикого родича сільськогосподарської рослини і вибрати гени стійкості, які нам потрібні: і ми можемо зробити це в рекордно короткі терміни. Раніше це був процес, який займав 10 або 15 років і був схожий на пошук голки в копиці сіна. Тепер ми можемо клонувати ці гени за лічені місяці і за тисячі фунтів замість мільйонів.” – говорить він.
Дослідження, опубліковане в журналі Nature Biotechnology, повідомляє, що технологія AgRenSeq була успішно випробувана на дикому родичі пшениці. Дослідники ідентифікували і клонували чотири гени стійкості до збудника стеблової іржі протягом місяця замість десяти років з допомогою звичайних засобів.
Роботу на дикій пшениці використовують як доказ концепції в цілому, розробляючи метод, який буде використовуватися для захисту багатьох культур, які мають диких родичів, включаючи сою, горох, бавовник, кукурудзу, картоплю, пшеницю, ячмінь, рис, банан і какао. Сучасні сільгоспкультури, створювані для високих врожаїв та інших бажаних агрономічних ознак, втратили велику генетичну різноманітність, особливо в сфері стійкості до хвороб.авовна, кукурудзу, картоплю, пшеницю, ячмінь, рис, банан і какао.
Повторне введення генів стійкості до хвороб від диких родичів є економічним і екологічно стійким підходом до розведення більш стійких культур. Однак інтрогреція цих генів у господарстві є трудомістким процесом з використанням традиційних методів селекції.
Новий метод поєднує в собі високопродуктивне секвенування ДНК з найсучаснішою біоінформатикою.
Це суміш двох технологій: генетики асоціації, і захоплення послідовності.
Для перевірки методу команда зібрала панель з 151 штамів трави під назвою Aegilops tauschii – прабатька сучасної пшениці. Цей дикий родич вніс основу D-геному в пшеницю.
Команда засіяла популяцію дикого родича патогеном стовбурної іржі і проаналізувала рослини для виявлення резистентних і сприйнятливих до захворювання механізмів. Зіставляючи цю інформацію з послідовностями ДНК рослин, вони змогли виявити ідентичність генів функціональної стійкості в популяції.
“Тепер у нас є “бібліотека” генів стійкості до хвороб, і ми розробили алгоритм, який дозволяє дослідникам швидко сканувати цю бібліотеку і знаходити гени функціональної стійкості” — вказав д-р Сану Арора, перший автор статті.
Лабораторія доктора Вульфа також є піонером в техніці швидкісної селекції, яка використовує покращене світлодіодне освітлення для прискорення генетичного поліпшення сільськогосподарських культур.
“AgRenSeq — це надійний протокол для швидкого виявлення генів стійкості.”
Інфоіндустрія за матеріалами farminguk.com
Читайте нас у TelegramПов’язані теми:





